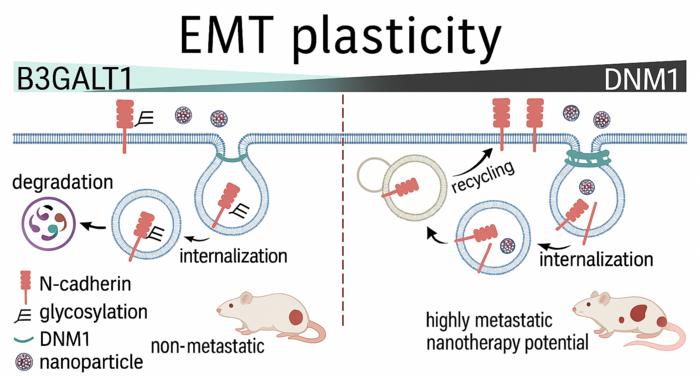
Now Reading: Tümörlerin Üç Boyutlu Analizi
-
01
Tümörlerin Üç Boyutlu Analizi
Tümörlerin Üç Boyutlu Analizi
Kanser araştırmalarında çığır açan yeni bir çalışma, tümör mikroçevresinin anlaşılma biçimini kökten değiştiriyor. Max Delbrück Moleküler Tıp Merkezi (MDC) bünyesinde Prof. Nikolaus Rajewsky liderliğinde yürütülen bu çalışma, Berlin Medikal Sistem Biyolojisi Enstitüsü’nün deney ve bilişim altyapısını bir araya getirerek, akciğer tümöründeki hücresel ekosistemlerin üç boyutlu haritasını oluşturuyor. Yenilikçi spatial transcriptomics (mekânsal transkriptomik) teknolojisi ile geliştirilen bu haritalama, tümörün sadece hücresel yapısını değil, hücreler arasındaki konumsal ilişkiler ve iletişim ağlarını da detaylıca ortaya koyarak kişiselleştirilmiş kanser tedavileri için yeni ufuklar açıyor.
Tümörler, artık sadece kötü huylu hücrelerin kümeleri olarak değil, kompleks ve dinamik biyolojik ekosistemler olarak değerlendiriliyor. Kanser hücreleri ile bağışıklık hücreleri, fibroblastlar ve çevresel ekstrasellüler matriks (ECM) arasında geçen karşılıklı etkileşimler hastalığın ilerlemesini ve tedaviye yanıtı doğrudan etkiliyor. Geleneksel patoloji yöntemleri ise çoğunlukla iki boyutlu doku kesitleri ve histolojik boyamalarla sınırlı kaldığından, mevcut yaklaşımlar tümör mikroçevresinin üç boyutlu ve moleküler dinamiklerini derinlemesine anlamakta yetersiz kalıyordu. Rajewsky ve ekibi, bunun üstesinden gelmek için yüksek çözünürlüklü tek hücre mekânsal teknolojilerini uygulayarak, tümör mimarisini moleküler ve hücresel komşuluk düzeyinde ayrıntılı şekilde ortaya koydu.
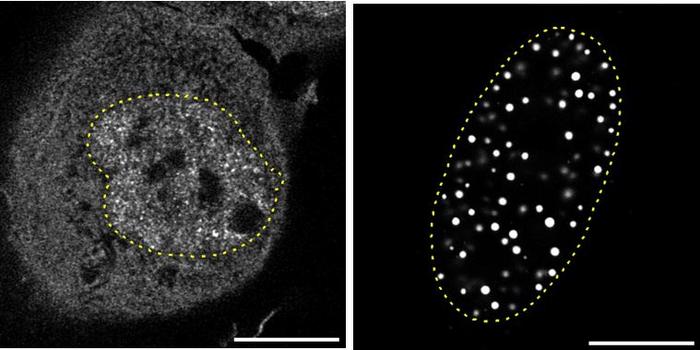
Mekânsal transkriptomik, RNA ifadesini moleküler detay ve uzamsal bağlam ile aynı anda ölçme olanağı tanıyan devrimsel bir teknoloji olarak öne çıkıyor. Geleneksel transkriptomik incelemeler, dokudaki RNA’yı homojenize ederek toplu analiz yaparken, mekânsal transkriptomik her hücrenin konumunu koruyarak transkript seviyelerini belirliyor. Rajewsky’nin ekibi, NanoString’in CosMx platformunu kullanarak, hücre başına 1.000’e kadar RNA molekülünü eşzamanlı olarak tespit edebildi. Bu, önceki yöntemlerin birkaç molekülden ibaret sınırlamalarının çok ötesinde olup, tek bir akciğer tümöründen 340 binden fazla hücrenin profillenmesini, 18 farklı hücre tipinin tanımlanmasını sağladı. Böylece tümörün heterojen yapısına dair eşi benzeri görülmemiş bir veri seti elde edildi.
Fakat iki boyutlu veri parçacıklarını üç boyutlu yapıya dönüştürmek, sadece deneysel değil çok ileri düzey hesaplama altyapısı gerektiriyordu. Araştırmacılar, STIM adlı özel algoritmayı geliştirerek, farklı mekânsal transkriptomik kesitleri üst üste bindiren ve hizalayan bir yöntemle sanal 3D doku blokları oluşturdu. Bu yaklaşımla, mekânsal veriler dijital görüntüler gibi işlenip, doku dilimleri 3D perspektifte tam anlamıyla birleştirildi. Bu inovatif çözüm, bilgisayarlı görü teknikleriyle biyoloji alanının kesiştiği nadir örneklerden oldu. Projede, Howard Hughes Tıp Enstitüsü’nden Dr. Stephan Preibisch’in bilişim uzmanlığı da sürece büyük katkı sağlayarak disiplinlerarası işbirliğinin önemini göstermiş oldu.
3D doku yeniden yapılandırmaları, sadece hücrelerin kimliklerini ortaya koymakla kalmadı, aynı zamanda ECM’nin yapısal bileşenlerini de detaylıca incelemeye imkân verdi. İkinci harmonik jenerasyon (SHG) mikroskopisi kullanılarak elastin ve kollajen lifleri gibi ECM elemanları görselleştirildi. Analizler, elastin açısından zengin bölgelerin sağlıklı doku ile ilişkili olduğunu; kollajen yoğun bölgelerin ise tümörün merkezinde ve çevresinde anormal doku remodelasyonuna işaret ettiğini ortaya koydu. Bu bulgular, ECM’deki yapısal değişimlerin kanser progresyonuna nasıl katkı sağladığını moleküler düzeyde açıklayan önemli kanıtlar sundu.
Daha da önemlisi, bu yöntem tümör içindeki hücresel etkileşimleri dinamik olarak gözler önüne serdi. Fibroblastlar, tümör büyümesini destekleyen bir doku iskeleti oluşturacak şekilde aktive oldu ve ECM’yi yeniden şekillendirdi. Sadece statik bir görüntü değil, işlevsel fenotipler ve hücreler arası sinyal iletimi haritalandı. Özellikle kanser hücrelerinin bağışıklık hücrelerinin dokuya sızmasını nasıl engellediğine dair mekanizmalar detaylandı. Tümör çekirdeğini çevreleyen bağışıklık nişleri var olmasına rağmen, bu bölgedeki bağışıklık hücrelerinin fonksiyonlarının tümör kaynaklı baskılanma nedeniyle kısıtlandığı tespit edildi.
Hücreler arası moleküler iletişimin kanserin bağışıklık sisteminden kaçışını nasıl sağladığına ilişkin bu detaylar, bağışıklık kontrol noktası (immune checkpoint) inhibisyonuna dayalı tedavilerin etkinliğini doğruladı. Tümörün neden olduğu lokal bağışıklık baskısını geri çevirebilen bu tedavi stratejileri, yoksa hareketsiz kalan bağışıklık hücrelerini açığa çıkararak daha hedefe yönelik ve etkili kişisel tedaviler sunabiliyor. Bu da klasik kemoterapilerin önüne geçen bir yaklaşımla hasta sonuçlarını iyileştirme potansiyeli taşıyor.
Çalışmanın dikkat çeken bir diğer yanı, kullanılan teknoloji ve yöntemlerin işleyişinin klinik rutinle uyumlu olması. MDC ekibi, klasik patoloji laboratuvarlarında sıklıkla tutulan formalin fikse edilmiş parafin blok (FFPE) doku kesitleri üzerinde bu yüksek çözünürlüklü analizlerin yapılabileceğini gösterdi. Bu “Patoloji 2.0” olarak adlandırılan yaklaşım, geleneksel mikroskobik incelemeyi moleküler ve mekânsal zenginliklerle genişletip, onkoloji tanı ve tedavi kararlarında devrim yaratacak kapasiteye işaret ediyor.
Araştırmanın pratik uygulama alanları ise çok geniş. Tümör içi hücresel mahalleler ve moleküler sinyal ağları haritalandığında, hekimler immünoterapi ve diğer tedavi yöntemlerini hastaya özgü olarak tasarlayabilecek. Daha büyük hasta gruplarında yürütülen analizlerle, moleküler hedeflerin doğrulanması ve kişiselleştirilmiş tedavi protokollerinin geliştirilmesi hedefleniyor. Bu şekilde, yaklaşımlar yalnızca teorik değil, etkili klinik araçlara evrilecek.
Bir sonraki aşamada çalışmaya proteomik verilerin entegrasyonu dâhil edilecek. MDC bünyesindeki Dr. Fabian Coscia’nın Spatial Proteomics Laboratuvarı ile yapılan işbirliği, RNA ekspresyonunun yanı sıra protein aktivite düzeylerinin de aynı tek hücre ve mekânsal bağlamda ölçülmesini sağlayacak. Bu çok-omik entegrasyon, tümör biyolojisinin işlevselliğini derinlemesine çözümleyip, post-transkripsiyonel düzenleme mekanizmalarını ve potansiyel tedavi hedeflerini daha net ortaya koyacak.
Sonuç olarak, bu çalışma kanser araştırması ve tanısında paradigma değişimine imza atıyor. Tek hücre çözünürlüğünde mekânsal transkriptomik, gelişmiş ECM görüntüleme teknikleri ve yenilikçi hesaplama metodolojisinin entegrasyonu sayesinde, tümör mikroçevresi moleküler ve mekânsal açıdan daha önce hiç olmadığı kadar şeffaf hale getirildi. Elde edilen veriler sadece hangi hücrelerin varlığını değil, nerede olduklarını, nasıl etkileştiklerini ve neden farklı fonksiyonlara sahip olduklarını ortaya koyuyor. Bunlar, tedavi planlamasında hastaya özgü ve daha etkili yaklaşımların geliştirilmesinde vazgeçilmez bilgiler.
Prof. Rajewsky bu gelişmeyi şöyle özetliyor: “Hasta tümörlerinden elde edilen kapsamlı veriler, kanser fenotiplerini belirleyen moleküler mekanizmaların bilgisayar destekli tahminlerini mümkün kılıyor.” Bu da onkolojide genel geçer tedavilerden bireye özel müdahalelere doğru dönüşümü tetikleyebilecek devrimci bir potansiyele sahip.
Biyoloji, bilişim bilimi ve gelişmiş görüntüleme tekniklerinin buluştuğu bu yeni çağda, yüksek teknolojili analiz araçları alışılmış patoloji tanılarını kişiselleştirilmiş kanser bakımına dönüştürebilecek güçlü bir platform olarak öne çıkıyor. Mekânsal, moleküler ve işlevsel verilerin entegrasyonu, yeni nesil tanı ve tedavi yöntemlerinin temelini oluştururken, akciğer kanseri ve diğer malignitelerle mücadelede hastalara umut vadediyor. Önümüzdeki on yılda bu yeniliklerin yaygınlaşarak kanser yönetim stratejilerini köklü biçimde değiştirmesi bekleniyor.
—
Araştırma Konusu:
Hücreler
Makale Başlığı:
Combining spatial transcriptomics and ECM imaging in 3D for mapping cellular interactions in the tumor microenvironment
Haberin Yayın Tarihi:
11 Nisan 2025
Web References:
10.1016/j.cels.2025.101261
Resim Credits:
Rajewsky laboratuvarı, Max Delbrück Merkezi
Anahtar Kelimeler:
spatial transcriptomics, 3D tumor mapping, extracellular matrix imaging, single-cell analysis, lung cancer, tumor microenvironment, immunotherapy, computational modeling, personalized medicine